Einführung
Willkommen bei Genestrip Web!
Genestrip Web ist eine Software für die Metagenomische Analyse von DNA-Sequenzierungsdaten. Über eine einfache Web-Oberfläche können Sie eine entspr. Analyse auch ohne Schulung oder spezielles IT-Fachwissen durchführen.
GeneStrip Web besteht aus drei Teilen:
- Die Web-basierte Benutzeroberfläche mit einem Programmteil zur Verarbeitung der DNA-Daten.
- Das eigentliche Analyseprogramm namens GeneStrip.
- Einer Sammlung von sogenannten Datenbanken, um DNA-Fragmente dem passenden Organismus zuzuweisen.
Genestrip Web benötigt Eingabedateien für die Analyse im fastq-Format - also als sogenannte fastq-Dateien. Diese müssen zuvor mit einem DNA-Sequencer aus einer Umwelt- oder Gewebeprobe erzeugt werden. Der Schritt ist aufwändig und kann meist nur von geschultem Personal in biologischen bzw. genetischen Fachlaboren durchgeführt werden. Die anschließende Analyse von fastq-Dateien mit GeneStrip Web ist hingegen einfach.
Das Ergebnis einer Analyse mit GeneStrip Web ist eine CSV-Datei, die man z.B. mit MS Excel öffnen kann oder auch direkt mit GeneStrip Web ansehen kann. Die Datei enthält verschiedene statistische Werte darüber, für welche Organismen “Treffer” in der fastq-Datei vorliegen. Daraus können Sie häufig Rückschlüsse über die in der Probe vorhandenen Organismen ziehen.
Die folgende Abbildung zeigt den schematisch gesamten Prozess.
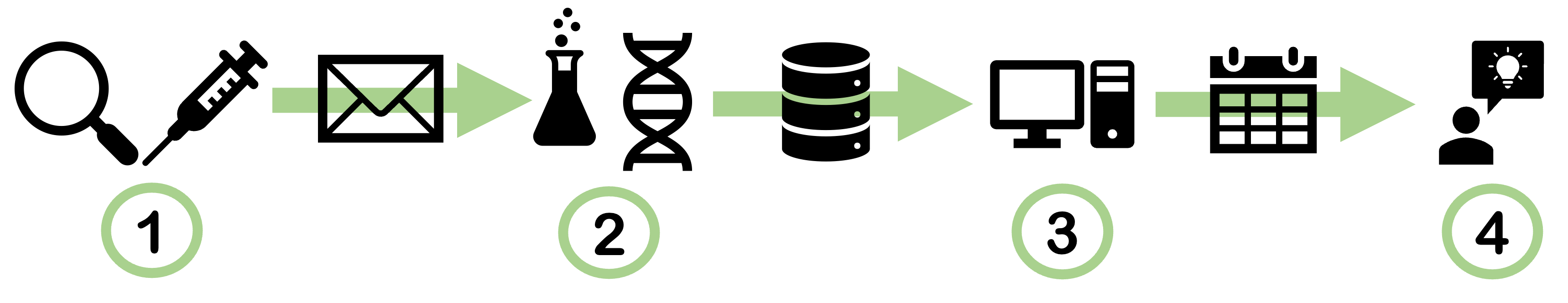
- Die Probenentnahme liefert eine Umwelt- oder Gewebeprobe.
- Die DNA-Sequenzierung der Probe liefert eine oder mehrere fastq-Dateien.
- Die Analyse mit GeneStrip-Web liefert dazu CSV-Dateien.
- Die CSV-Dateien werden von Forschern und Experten genutzt, um Rückschlüsse über die in der Probe vorhandenen Organismen zu ziehen.
Wichtig: Das Analyseverfahren mit Genestrip Web bzw. Genestrip ist keinesfalls für die medizinische Diagnostik geeeignet!
Online-Demo
Eine Online-Demo von Genestrip Web finden Sie hier:
https://genestrip.it.hs-heilbronn.de/gweb
Die Online-Demo zeigt u.a. Analyseergebnisse zur Analyse von 6 Zecken entsprechend der Nature-Publikation “Metagenomic surveillance for bacterial tick-borne pathogens using nanopore adaptive sampling”.
Dazu wurden die fastq-Dateien aus der Publikation mit Genestrip Web analysiert. Es konnten alle ensprechenden Ergebnisse aus der Publikation nachvollzogen werden. Sie können diese ohne Anmeldung in der Online-Demo ansehen. Auch Informationen zu einigen der bereitgestellten Datenbanken können Sie ohne Anmeldung in der Online-Demo ansehen.
Analysen können in der Online-Demo nicht ohne Anmeldung durchgeführt werden.
Vorteile im Überblick
- GeneStrip Web kann mit wenigen Klicks auf gängigen, modernen Windows-PCs installiert und betrieben werden, während andere metagenomische Analysewerkzeuge vornehmlich auf Unix-Systemen arbeiten. (Genestrip Web kann zusätzlich auch auf Macs, Linux usw. betrieben werden.)
- GeneStrip Web stellt eine einfach zu bedienende Web-Oberfläche bereit, während andere metagenomische Analysewerkzeuge über Kommandozeilenbefehle bedient werden müssen. (Das enthaltene Kernsystem GeneStrip kann dennoch ebenso von der Kommendozeile aus benutzt werden.)
- GeneStrip Web stellt eine interessante und praxis-relevante Sammlung von Datenbanken zur einfachen Installation bereit, die viele menschliche Pathogene abdeckt. Weitere Datenbanken können von IT-Administoren mit moderatem Aufwand mittels GeneStrip generiert werden.
- Im Vergleich zu alternativen Systemen, wie etwa KrakenUniq, benötigt GeneStrip Web vergleichsweise wenig Hauptspeicher für entsprechend spezialisierte Analysedatenbanken.
- Im Gegensatz etwa zu Kraken2 liefert GeneStrip Web sehr exakte und umfangreiche Analyseergebnisse für zugeordnete Organismen.
Lizenz
Genestrip Web oder kurz “gweb” ist frei für den nicht-kommerziellen Gebrauch unter dieser Lizenz (in englisch).
 Genestrip Web Hilfeseiten
Genestrip Web Hilfeseiten Hochschule Heilbronn
Hochschule Heilbronn